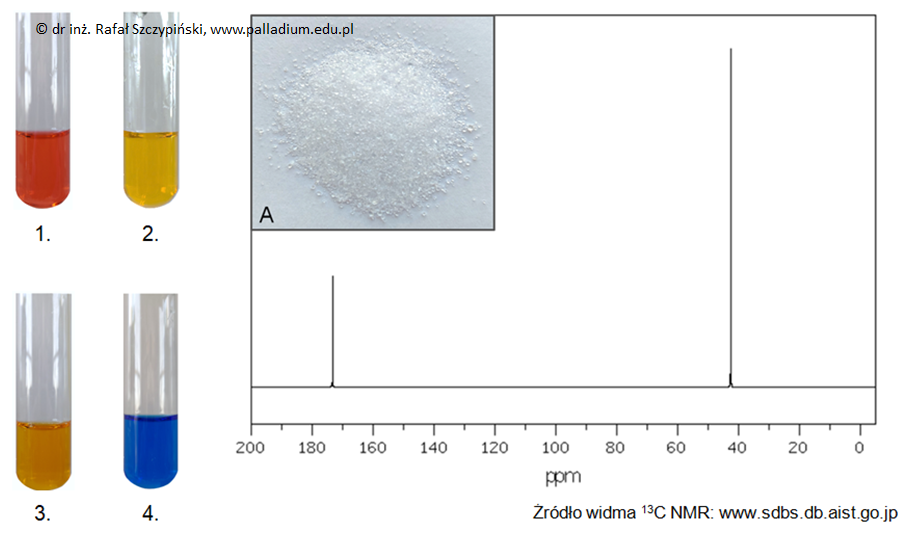
Zadanie 29.1. Wybór właściwego odczynnika oraz wskazanie nazwy powstałej barwnej substancji. Doświadczenia chemiczne w zadaniach część 1. Rozdział 5 (1 punkt)
Przeprowadzono eksperyment z wykorzystaniem materiału celulozowego (fotografia A) oraz odczynnika X według zamieszczonego schematu.
HCl(aq), CuO(s), Br2(c), H2SO4(stężony).

Wskaż wykorzystany odczynnik i podaj nazwę barwnej substancji powstałej w efekcie jego działania. Wybrany odczynnik: Nazwa substancji: © dr inż. Rafał Szczypiński, wszelkie prawa zastrzeżone
Zadanie 29.2. Zapis nazwy własności wykazywanej przez wybrany odczynnik. Doświadczenia chemiczne w zadaniach część 1. Rozdział 5 (1 punkt)
Przeprowadzono eksperyment z wykorzystaniem materiału celulozowego (fotografia A) oraz odczynnika X według zamieszczonego schematu.
HCl(aq), CuO(s), Br2(c), H2SO4(stężony).

Podaj nazwę własności jaką wykazuje wybrany odczynnik podczas tego eksperymentu. © dr inż. Rafał Szczypiński, wszelkie prawa zastrzeżone
Zadanie 30. Przypisywanie fotografii do odpowiedniej substancji z uzasadnieniem. Doświadczenia chemiczne w zadaniach część 1. Rozdział 5 (1 punkt)
Ksylitol jest polihydroksylowym alkoholem, białym ciałem stałym o wzorze sumarycznym C5H7(OH)5. W jego cząsteczce każdy atom węgla połączony jest z jedną grupą funkcyjną. W temperaturze 22 oC związek ten bardzo dobrze rozpuszcza się w wodzie.
Do probówek zawierających ciepłą wodę destylowaną wprowadzono próbki trzech substancji chemicznych – sacharozy, skrobi ziemniaczanej oraz ksylitolu. Zawartością każdego z naczyń energicznie wstrząśnięto i pozostawiono na kilkadziesiąt sekund. Ponieważ wygląd dwóch spośród trzech uzyskanych mieszanin był praktycznie identyczny, wykonano jedynie dwie fotografie, które oznaczono literami A oraz B.

Napisz oznaczenie fotografii, która przedstawia mieszaninę uzyskaną po wprowadzeniu do wody destylowanej danej substancji chemicznej – uzupełnij puste pola. Uzasadnij swój wybór. sacharoza: skrobia: ksylitol: Uzasadnienie: © dr inż. Rafał Szczypiński, wszelkie prawa zastrzeżone
Zadanie 32. Zapis nazwy wybranego odczynnika oraz wskazanie numeru probówki. Doświadczenia chemiczne w zadaniach część 1. Rozdział 5 (1 punkt)
Ksylitol jest polihydroksylowym alkoholem, białym ciałem stałym o wzorze sumarycznym C5H7(OH)5. W jego cząsteczce każdy atom węgla połączony jest z jedną grupą funkcyjną. W temperaturze 22 oC związek ten bardzo dobrze rozpuszcza się w wodzie.
Do probówek zawierających ciepłą wodę destylowaną wprowadzono próbki trzech substancji chemicznych – sacharozy, skrobi ziemniaczanej oraz ksylitolu. Zawartością każdego z naczyń energicznie wstrząśnięto i pozostawiono na kilkadziesiąt sekund. Ponieważ wygląd dwóch spośród trzech uzyskanych mieszanin był praktycznie identyczny, wykonano jedynie dwie fotografie, które oznaczono literami A oraz B.

W celu wykazania, że w jednej z probówek znajdowała się skrobia ziemniaczana przeprowadzono doświadczenie chemiczne wybierając jeden spośród odczynników:
HCl(aq), odczynnik Tollensa, płyn Lugola, Br2(c), H2SO4(stężony).
Jedna z poniższych fotografii przedstawia zawartość probówki uzyskaną po wprowadzeniu do niej jednej z wymienionych wyżej substancji.

Napisz nazwę wybranego odczynnika oraz wskaż numer fotografii przedstawiającej naczynie zawierające skrobię przed rozpoczęciem doświadczenia. Nazwa odczynnika: Numer probówki: © dr inż. Rafał Szczypiński, wszelkie prawa zastrzeżone
Zadanie 33.1. Wskazanie numeru probówki z pozytywnym wynikiem próby biuretowej. Doświadczenia chemiczne w zadaniach część 1. Rozdział 5 (1 punkt)
Fotografie 1.–4. przedstawiają albuminę (białko jaja kurzego) poddaną procesowi denaturacji przy użyciu odczynników:
H2SO4(stężony), CuSO4(aq), HNO3(stężony), NaOH(stężony).

Wskaż numer tej probówki, której zawartość przedstawia pozytywny wynik próby biuretowej. Napisz wzory sumaryczne związków chemicznych, których roztwory wykorzystano. Numer probówki: Wzory sumaryczne: © dr inż. Rafał Szczypiński, wszelkie prawa zastrzeżone
Zadanie 33.2. Ocena z uzasadnieniem możliwości obecności reszty tyrozyny lub tryptofanu w białku. Doświadczenia chemiczne w zadaniach część 1. Rozdział 5 (1 punkt)
Fotografie 1.–4. przedstawiają albuminę (białko jaja kurzego) poddaną procesowi denaturacji przy użyciu odczynników:
H2SO4(stężony), CuSO4(aq), HNO3(stężony), NaOH(stężony).

Oceń, czy wśród aminokwasów, których reszty budują łańcuchy polipeptydowe albuminy może znajdować się tyrozyna lub tryptofan? Uzasadnij odpowiedź – odnieś się do wyglądu zawartości odpowiedniego naczynia. Ocena: Uzasadnienie: © dr inż. Rafał Szczypiński, wszelkie prawa zastrzeżone
Zadanie 33.3. Wskazanie numeru właściwej probówki z zawartością. Doświadczenia chemiczne w zadaniach część 1. Rozdział 5 (1 punkt)
Fotografie 1.–4. przedstawiają albuminę (białko jaja kurzego) poddaną procesowi denaturacji przy użyciu odczynników:
H2SO4(stężony), CuSO4(aq), HNO3(stężony), NaOH(stężony).

Wskaż numer tej probówki, w której za denaturację odpowiada wyłącznie obecność kationów metalu ciężkiego. © dr inż. Rafał Szczypiński, wszelkie prawa zastrzeżone
Zadanie 34. Wskazanie numeru właściwej probówki oraz oznaczenia próbki furanozy. Doświadczenia chemiczne w zadaniach część 1. Rozdział 5 (1 punkt)
Rysunki A i B przedstawiają (odpowiednio) wzory Hawortha dwóch cukrów – β-D-ksylulofuranozy oraz β-D-arabinofuranozy. W celu ich odróżnienia zaproponowano zestawy odczynników:
KMnO4(aq), odczynnik Tollensa, odczynnik Trommera, Br2(aq) z dodatkiem KHCO3.
Fotografie 1.–4. ilustrują efekt doświadczenia przeprowadzonego z udziałem jednego z wymienionych monosacharydów oraz każdego z podanych odczynników.
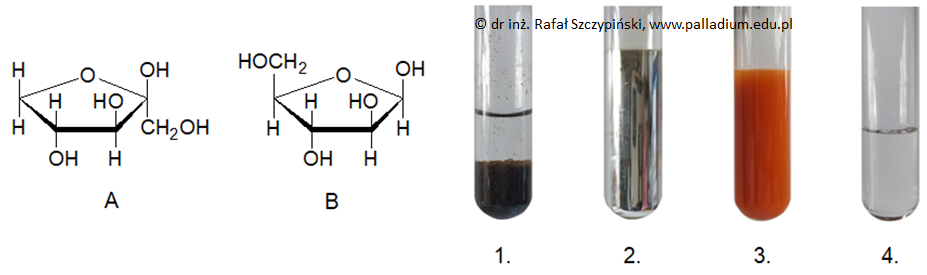
Podaj oznaczenie literowe próbki tej furanozy, która została wykorzystana podczas doświadczenia. Wskaż numer probówki potwierdzającej użycie wskazanego cukru. Oznaczenie literowe: Numer probówki: © dr inż. Rafał Szczypiński, wszelkie prawa zastrzeżone
Uzupełnienie schematu klatkowego dla stanu wzbudzonego. Zadanie 1.1. Arkusz CKE chemia próbny grudzień 2024 rozszerzony (1 punkt)
Atomy pierwiastków chemicznych mogą występować w różnych stanach energetycznych. Stan o najniższej energii nazywa się stanem podstawowym, a stany o wyższych energiach to stany wzbudzone. Atom X w stanie podstawowym ma elektrony rozmieszczone na trzech powłokach. Poza tym wiadomo, że dwie podpowłoki należące do zewnętrznej powłoki nie mają pustych orbitali, a jedna z nich zawiera dwa niesparowane elektrony.
W pewnych warunkach atom X w stanie podstawowym pochłonął energię. Nastąpiła zmiana stanu energetycznego tylko jednego elektronu, co spowodowało wzrost liczby niesparowanych elektronów w tym atomie. Przed wzbudzeniem stan tego elektronu był opisywany wartościami liczb kwantowych 𝑛 = 3, 𝑙 = 1, a po wzbudzeniu zmieniła się tylko wartość liczby pobocznej (orbitalnej).
Uzupełnij poniższy schemat, tak aby przedstawiał on klatkowy zapis konfiguracji elektronowej atomu X w opisanym stanie wzbudzonym.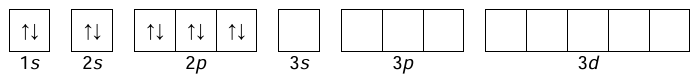
Określenie symbolu pierwiastka oraz położenia w układzie okresowym. Zadanie 1.2. Arkusz CKE chemia próbny grudzień 2024 rozszerzony (2 punkty)
Atomy pierwiastków chemicznych mogą występować w różnych stanach energetycznych. Stan o najniższej energii nazywa się stanem podstawowym, a stany o wyższych energiach to stany wzbudzone. Atom X w stanie podstawowym ma elektrony rozmieszczone na trzech powłokach. Poza tym wiadomo, że dwie podpowłoki należące do zewnętrznej powłoki nie mają pustych orbitali, a jedna z nich zawiera dwa niesparowane elektrony.
Uzupełnij poniższą tabelę. Wpisz symbol pierwiastka X, numer okresu i numer grupy w układzie okresowym oraz symbol bloku konfiguracyjnego, do którego należy pierwiastek X.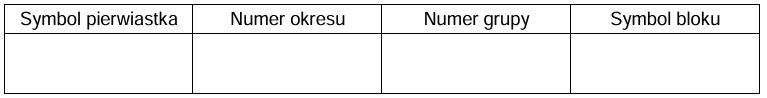
Przypisywanie liczb kwantowych orbitalom. Zadanie 2. Arkusz CKE chemia próbny grudzień 2024 rozszerzony (1 punkt)
Na poniższym rysunku przedstawiono kontury dwóch orbitali atomowych A i B należących do tej samej podpowłoki elektronowej.
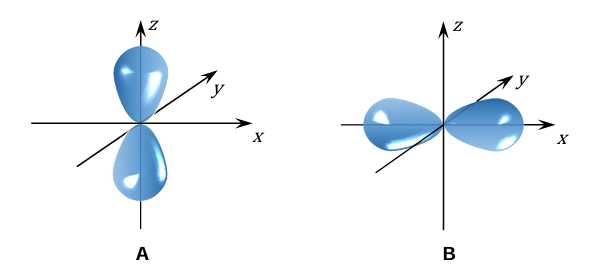
Uzupełnij zdania dotyczące przedstawionych na rysunku orbitali atomowych A i B. Najmniejsza wartość głównej liczby kwantowej 𝑛, która może opisywać każdy z przedstawionych orbitali, wynosi: . Orbital A i orbital B różnią się wartością liczby kwantowej oznaczonej symbolem .